Metagenômica Análises – Diversidade bacteriana afeta expressivamente os níveis de amônia e gerais de nitrogênio em Sistemas Aquabioponics bioflocs, baseados no 16S rRNA Gene Amplicon Metagenomics**
Aecio D’Silva 1 , John A Kyndt 2 *
1 Departamento Moura Enterprises, Divisão de Biotecnologia da Aquicultura, Tucson, AZ 85742, EUA; 2 Departamento de Faculdade de Ciências e Tecnologia, Universidade de Bellevue, Bellevue, NE 68005, EUA
**Pesquisa publicada originalmente no Applied Microbiology Journal Volume 6, Issue 1(2020)
RESUMO
Metagenômica Análises – O sistema AquaBioPonics Bioflocs, que combina sinergicamente a aquicultura com a tecnologia dos bioflocs, cultivo de algas, hidroponia e maximização do uso d’água, é uma biotecnologia eficiente na aquicultura para a produção de peixes, camarões e orgânicos legumes. Ao adicionar um probiótico comercial a nossos sistemas, observamos uma produtividade aprimorada e controle eficiente (concentrações mais baixas) de amônia. Nesta publicação, damos informações iniciais sobre o microbioma em geral e a presença de bactérias metabolizantes de nitrogênio presentes nesses sistemas de aquicultura de alto rendimento.
Palavras-chave: Aquicultura; Metagenômica Análises; Bactérias Comammox; Nitrospira ; Probióticos; AquaBioPonics Bioflocs
INTRODUÇÃO
A eficiência dos sistemas AquaBioPonics pode ser fortemente aprimorada quando a tecnologia de bioflocos (BFT) é adicionada como técnica de gestão da qualidade da água. Os bioflocos, por sua definição, são agregações agrupadas de comunidades microbianas, como fitoplâncton, zooplâncton, bactérias matéria orgânica viva e morta articulada. Camarão e tilápia beneficiam especialmente da BFT devido à sua capacidade de filtrar os flocos na coluna d’água, reduzindo assim os custos de alimentação, melhorando de conversão alimentar [1,2]. Recentemente, estudos também mostraram que os bioflocs podem ser coletados de sistemas de cultura, secados e adicionados como componente na ração granulada para aquicultura, substituindo 2/3 da farinha de peixe e 100% das farinhas vegetais. Contudo, a viabilidade econômica de empregar flocos como componente de ração seca exige mais pesquisa [3].
Para promover a produção de bioflocos, é importante controlar a pH (7-8), amônia, nitrito, nitrato e lodo nos tanques/raceways. Nos sistemas de bioflocos, existem três processos principais de controle de amônia: captação pelas algas, assimilação bacteriana e nitrificação [4]. Para maximizar esses processos, equilibrando a relação C:N é a principal estratégia de gerenciamento do controle de amônia em sistemas de bioflocos, entretanto, espera-se que a diversidade bacteriana presente na aquicultura desempenhe também um papel muito importante. Para estudar a diversidade bacteriana, testamos nossos Sistemas AquaBioPonic Bioflocs com probióticos (APB; Aquabioponics ProBiotics) e sem a aplicação comercial de probióticos (ANPB; Aquabioponics Non-ProBiotics). Estas fórmulas de probióticas são projetadas para digerir o lodo orgânico, melhorar a FCR e reduzir amônia, nitrito e nitrato. Neste trabalho de Metagenômica Análises, descrevemos como a concentração do amônio nas culturas foram reduzidas a níveis muito baixos, indicando que o amônio foi transformado em biomassa microbiana. Para entender mais sobre os processos microbianos envolvidos, comparamos a composição microbiana usando o 16S RNA amplicon metagenomics entre tanques com (APB) e sem adições de probióticos (ANPB).
MATERIALS E MÉTODOS
Os experimentos duraram 60 dias na ACI FishFarm, em Florença Arizona, Estados Unidos usando 6 tanques circulares de alevinagem (10 metros cúbicos – 12.000 peixes com peso médio inicial de 0,9 g – peso médio final de 32,7 g) e 6 raceways (201 metros cúbicos com 12.000 peixes com peso médio inicial de 36,5 g – peso médio final 153,5g), comparando-os com outros tanques / raceways também em plena produção, mas sem o uso do probiótico (Figura 1). Os peixes (tilápias) estavam já por 15 dias nos tanques berçários circulares e 35 dias nos raceways, antes de aplicar os probióticos em uma concentração final de 0,5 mg / L. As concentrações totais de amônio (expressas em NH3-N), e as concentrações de nitrito e nitrato foram medidas usando o kit Hach de reagente total de nitrogênio, nitrito e nitrato (Hach).
Dados dos Pesquisadores
Correspondência para: John A Kyndt, Departamento de Faculdade de Ciência e Tecnologia, Universidade de Bellevue, Bellevue, NE 68005, EUA, E-mail:jkyndt@bellevue.edu
ou Aecio D’Silva, Moura Biotechnology Division, E-mail: support@mybelojardim.com
Recebido: 20 de junho de 2020; Aceito: 4 de julho de 2020; Publicado: 10 de julho de 2020
Citação: D’Silva A, Kyndt JA (2020) Diversidade bacteriana afeta expressivamente os níveis de amônia e gerais de nitrogênio em Sistemas Aquabioponics bioflocos, Baseado em 16S rRNA Gene Amplicon Metagenomics. Applied Microbiol Open Access 6: 169. Doi: 10.35248 / 2471-9315.20.6.169
Direitos autorais: © 2020 D’Silva A, et al. Este é um artigo de acesso aberto distribuído sob os termos da Creative Commons Attribution License, que permite uso irrestrito, distribuição e reprodução em qualquer meio, desde que o autor e a fonte originais sejam creditados.
Figura 1: Sistemas AquaBioPonics Bioflocs na ACI FishFarm, Florence Arizona, são configuradas como raceways de recirculação (imagens superiores) ou tanques berçários circulares (imagens inferiores).
Amostras de água para Metagenômica Análises 16S rRNA metagenomics foram coletadas em Setembro de 2019 dos nossos tanques e raceways, enviados durante a noite para o laboratório e armazenado a 4 ° C. Foram coletadas amostras de ambos os Tanques da ANPB e APB 10 dias após a adição dos probióticos nos tanques APB. As amostras foram coletadas em triplicata e agrupadas para cada condição. O DNA total foi extraído usando o PureLink Kit de purificação de DNA de microbioma (Invitrogen), resultando em uma absorvência 260/280 de 1,98 (ANPB) e 1,92 (APB) como medido por Nanodrop (Thermo Scientific). Um rRNA 16S biblioteca de sequenciamento de amplicons foi preparada para cada amostra, após a preparação da biblioteca de sequenciação metagenômica 16S (Illumina), usando iniciadores de amplicons direcionados ao V3 e regiões V4 [5]. As amostras foram sequenciadas usando um 1,8 pM biblioteca com um Illumina MiniSeq usando extremidade emparelhada de sequenciamento (2 × 150 bp). As sequências iniciadoras foram removidas e as leituras com baixo índices de qualidade (pontuação média <20) foram filtrados usando o Kit de ferramentas FASTQ (Illumina, versão 2.2.0). Os 16S O aplicativo Metagenomics no BaseSpace (versão 1.0.1) foi usado para classificação taxonômica, que utiliza um sistema de iluminação versão com curadoria do banco de dados taxonômicos GreenGenes [6]. Parâmetros padrão foram utilizados para todos os aplicativos de software.
RESULTADOS E DISCUSSÃO
Após 10-20 dias, a concentração total de amônio nos tanques e raceways com probióticos adicionados (APB) estavam constantemente em níveis muito baixos (= <1,0 ppm) indicando que o amônio foi transformado em biomassa microbiana. Nitrito e nitrato, e lodo também estavam todos dentro de níveis seguros. Antes de começarmos a usar probióticos e nos raceways / tanques ANPB, esses níveis atingiram 8,0 ppm em média e frequentemente aumentavam. Para controlar esses parâmetros, anteriormente tínhamos que aplicar constantemente uma mistura local de bioflocs (açúcares demerara / branco, melaço, amido, mandioca e farinhas de trigo, aveia, milho). Isso substancialmente aumentava a intensidade do trabalho para manter a boa qualidade da água. As diferenças básicas observadas na produção de peixes (tilápia) foram taxas de sobrevivência aumentadas (entre 95% e 99%) e taxa de conversão alimentar (= <1.0). Colhemos os peixes nos tanques (no final do experimento de 60 dias) e continuaram nos raceways até os peixes atingirem 850 g (tempo total: 135 dias).
Nestas Metagenômicas Análises, o DNA total foi extraído de amostras de água (5 ml) retiradas de tanques e raceways, obtendo um rendimento médio de 5 ng / ul para as amostras ANPB e 50 ng / ul para as amostras APB, com uma relação 260/280 de 1,98 (ANPB) e 1,92 (APB). O sequenciamento Illumina gerou 438.278 leituras para ANPB e 437.626 leituras para APB, que foram analisadas quanto à composição metagenômica usando o aplicativo Metagenomics no Basespace (Illumina). As duas amostras mostraram uma clara diferença de composição bacteriana em nível familiar e além. A Tabela 1 desta Metagenômica Análises ilustra as OTUs mais abundantes, mostrando uma significativa diferença ao nível da família entre as duas amostras. 79% das leituras foram classificadas de acordo com o gênero.
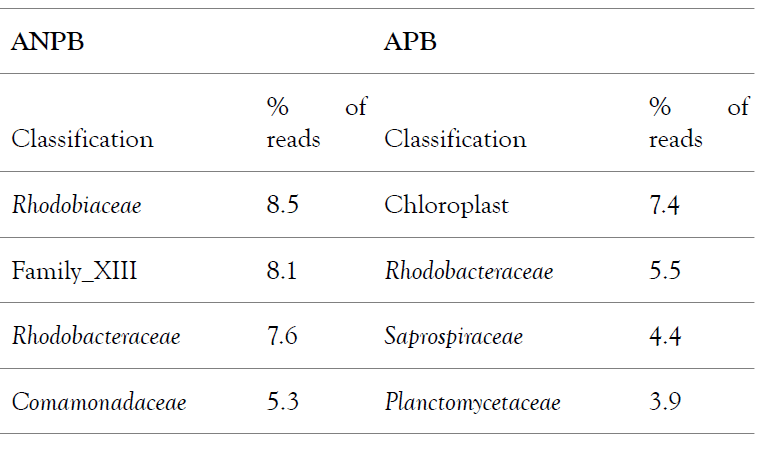
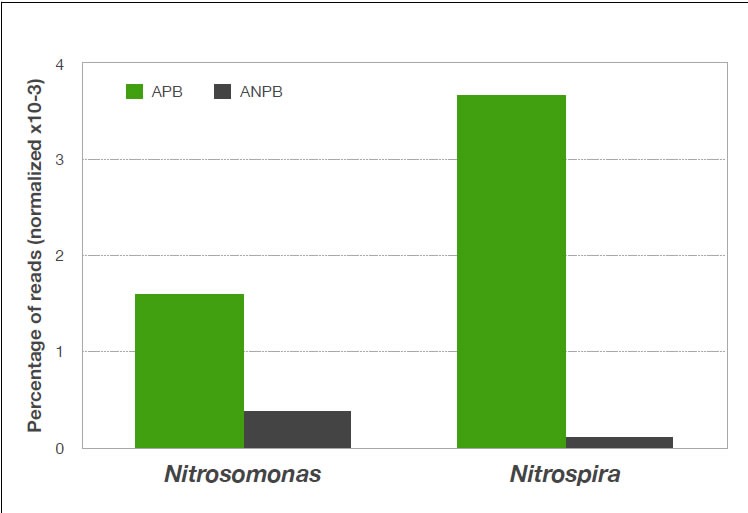
Notavelmente, não houve representação de cloroplasto nos melhores resultados dos ANPB, indicando menor crescimento de algas nessas culturas. De acordo com uma análise de diversidade de espécies de Shannon [7,8], cada uma das amostras continha cerca de 2000 espécies em potencial. Isto parece ser uma representação substancialmente mais alta de metabolizantes espécies na cultura APB em comparação com as culturas ANPB (Figura 2). Nitrosomonas continham 700 leituras no APB e167 leituras na ANPB e Nitrospiras continham 1608 leituras na APB contra apenas 48 leituras em espécies da ANPB. Nenhuma quantidade significativa das espécies Nitrobacter foram encontradas em ambas as amostras. Estes resultados são consistentes com nossa observação de significativamente menos amônia e maior assimilação de nitrogênio em nossas culturas APB.
Figura 2: Visão geral do gráfico das espécies metabolizadoras de nitrogênio nas culturas de APB comparadas às culturas de ANPB. Valores são apresentados como porcentagem de leituras para cada espécie, normalizada para o total número de leituras para cada amostra.
Vários isolados de Nitrospira são considerados comammox (COMpleteBactérias AMMonia OXidiser) [9,10] e estas parecem ser as principais espécies metabolizadoras de nitrogênio no sistema. Com base em nossa Identificação do 16SrRNA , Nitrospira japonica (NR_114396.1) foi a espécie dominantes de Nitrospira em nossas culturas, enquanto Nitrosomonas aestuarii (FR828476), Nitrosomonas oligotropha (FR828478) e Nitrosomonas ureae (FR828472) foram as principais espécies de Nitrosomonas . A nitrificação é uma parte central do ciclo do nitrogênio e, por mais de um século, a nitrificação foi pensado para ser um processo de duas etapas envolvendo oxidação de amônia para Nitrito por quimiolitoautotrófico microrganismos oxidantes de amônia, e oxidação de nitrito em nitrato por bactérias oxidantes de nitrito. Todos os comammox conhecidos são membros da género Nitrospira e possuem enzimas complementares para ambas funções de oxidação de amônia e nitrito [9,10]. Metagenômica evidência da presença de comammox em sistemas de distribuição de água potável [11, 12], filtros rápidos de areia alimentados por água subterrânea [13,14], processos de tratamento de águas residuais [15-18] e em solos de agricultura [19] foram relatados. Identificação de espécies com base apenas em análises do 16S rRNA é certamente limitante e foi demonstrado que o 16S rRNA sozinho não deve ser usado para identificar comammox no nível das espécies, porque essas bactérias não são distinguíveis através de análises filogenéticas rigorosas de bactérias oxidantes de nitrito do gênero Nitrospira . Seria necessária uma análise mais aprofundada para identificar a verdadeira identidade da espécie entre as dominantes espécies Nitrospira não cultivadas em nossos sistemas aquabiopônicos.
CONCLUSÃO
Os sistemas AquaBioPonics bioflocs demonstraram ser altamente eficientes, especialmente para a aquicultura de tilápia de alta densidade, e com base nessa análise, podemos concluir que a composição microbiana, particularmente bactérias comammox, parece desempenhar um papel importante na remoção do excesso de nitrogênio que envenenaria os peixes. Este método não apenas melhora a qualidade do nitrogênio da água, mas também reduz significativamente o trabalho envolvido na manutenção desses sistemas.
Disponibilidade de Dados
Os conjuntos de dados de 16S rRNA gene amplicon foram depositados em DDBJ / ENA / GenBank no projeto PRJNA599405 e pode ser acessado com os números de acesso SRA SRR10843757 (ANPB) e SRR10843772 (APB).
RECONHECIMENTOS
Este trabalho foi patrocinado pelo Wilson Enhancement Fund para pesquisa aplicada em ciências na Universidade de Bellevue. Indústrias do Complexo Correcional do Arizona (ACI) da Fazenda de Peixes em Florence, Arizona forneceram a facilidade para executar os experimentos. Dr. Marcos Eberlin da Universidade Presbiteriana Mackenzie , São Paulo, Brasil, por introduzir os autores às espécies Nitrospira.[20]
REFERÊNCIAS
1. McGraw B. Biofloc systems viable for tilapia production. Global Aquaculture Alliance. 2019;
2. Crab R, Defoirdt T, Bossier P, Verstraete W. Biofloc technology in aquaculture: Beneficial effects and future challenges. Aquaculture 2012; 351-356.
3. Hargreaves JA. Biofloc Production Systems for Aquaculture. SRAC Publication. No. 4503 2013;
4. Klindworth A, Pruesse E, Schweer, T, Peplies, J, Quast, C, Horn, M, et al. Evaluation of general 16S ribosomal RNA gene PCR primers for classical and next-generation sequencing-based diversity studies. Nucleic Acids Res. 2013; 41:1: e1.
5. Wang Q, Garrity, GM, Tiedje, JM, Cole, JR. Naïve Bayesian Classifier for Rapid Assignment of rRNA Sequences into the New Bacterial Taxonomy, Appl. Env. Microbiol. 2007; Vol. 73. 16:5261–5267.
6. Spellerberg IF, and Peter JF. A tribute to Claude Shannon and a plea for more rigorous use of species richness, species diversity, and the ‘Shannon–Wiener’ Index. Global Ecology and Biogeography. 2003; 12:3: 177-179.
7. Tuomisto H. A diversity of beta diversities: straightening up a concept gone awry. Part 1. Defining beta diversity as a function of alpha and gamma diversity. Ecography. 2010; 33: 2–22.
8. Daims H, Lebedeva E V, Pjevac P, Han P, Herbold C, Albertsen M, et al. Complete nitrification by Nitrospira bacteria. Nature. 2015; 528: 504–509.
9. Van Kessel MAHJ, Speth DR, Albertsen M, Nielsen PH, Op den Camp HJM, Kartal B, et al. Complete nitrification by a single microorganism. Nature. 2015; 528: 555–559.
10. Pinto AJ, Marcus DN, Ijaz UZ, Santos QMBD, Dick GJ, Raskin L. Metagenomic evidence for the presence of comammox Nitrospiralike bacteria in a drinking water system. mSphere. 2016;1:e00054-15.
11. Wang YL, Ma LP, Mao YP, Jiang XT, Xia Y, Yu K, et al. Comammox in drinking water systems. Water Res. 2017; 116:332– 341.
12. Bartelme RP, McLellan SL, Newton RJ. Freshwater recirculating aquaculture system operations drive biofilter bacterial community shifts around a stable nitrifying consortium of ammonia-oxidizing archaea and comammox Nitrospira. Front Microbiol. 2017; 8:101.
13. Fowler SJ, Palomo A, Dechesne A, Mines PD, Smets BF. Comammox Nitrospira are abundant ammonia oxidizers in diverse groundwater-fed rapid sand filter communities. Environ. Microbiol. 2018; 20:1002–1015.
14. Annavajhala MK, Kapoor V, Santo-Domingo J, Chandran K. Comammox functionality identified in diverse engineered biological wastewater treatment systems. Environ. Sci. Technol. Lett. 2018; 5:110–116.
15. Camejo PY, Domingo JS, McMahon KD, Noguera DR. Genomeenabled insights into the ecophysiology of the comammox bacterium “ Candidatus Nitrospira nitrosa. ” mSystems. 2017;2:e00059-17.
16. Fan XY, Gao JF, Pan KL, Li DC, Dai HH. Temporal dynamics of bacterial communities and predicted nitrogen metabolism genes in a full-scale wastewater treatment plant. RSC Adv. 2017; 7:56317–56327.
17. Chao Y, Mao Y, Yu K, Zhang T. Novel nitrifiers and comammox in a full-scale hybrid biofilm and activated sludge reactor revealed by metagenomic approach. Appl. Microbiol. Biotechnol. 2016;100:8225–8237.
18. Orellana LH, Chee-Sanford JC, Sanford RA, Loffler FE, Konstantinidis KT. Year-round shotgun metagenomes reveal stable microbial communities in agricultural soils and novel ammonia oxidizers responding to fertilization. Appl. Environ. Microbiol. 2018; 84:e01646.
19. Daims H, Lucker S, Wagner M. A new perspective on microbes formerly known as nitrite-oxidizing bacteria. Trends Microbiol. 2016; 24 : 699–712
20. Eberlin Marcos. Foresight: How the Chemistry of Life Reveals Planning and Purpose. 2019. Discovery Institute Press, Seattle, WA, USA.